5.7 调整数据 EMP_modify_assay
通常情况下,模块EMP_identify_assay能有效处理实验数据。然而在某些特殊情况下,需要手动调整数据以解决问题。
5.7.1 调整基因组数据
在基因表达数据中,有时会出现某些特征的表达量全部为零的情况,这可能会导致一些问题。为了尽可能保留信息,有时会将这些零值替换为一个极小的非零值。这样可以在一定程度上保持数据的结构,同时避免数值计算中的错误。
🏷️示例1:使用模块EMP_assay_extract将组学项目geno_ec的assay中的零值替换为伪计数0.0001。
调整数据前:
MAE |>
EMP_assay_extract('geno_ec')

调整数据后:
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_modify_assay('==0',pseudocount=0.0001)

🏷️示例2:使用模块EMP_modify_assay将组学项目geno_ec的assay中小于阈值10的绝对丰度值转换为伪计数5。
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_modify_assay('<10',pseudocount=5)

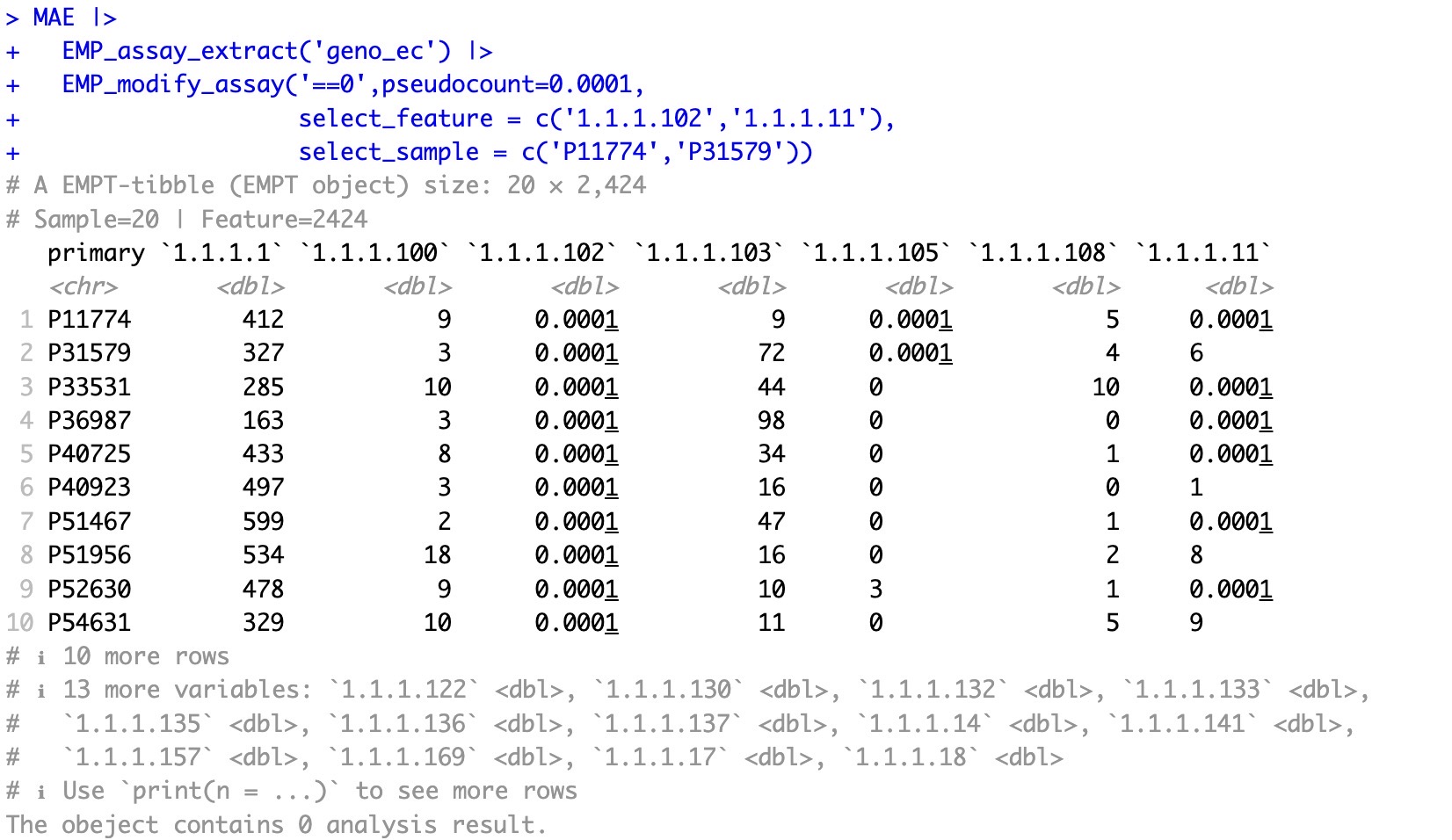
🏷️示例3:选择特定的样本和特征进行数据调整。
注意:
参数
参数
select_feature和参数select_sample可以针对特定样本和特征的数据进行调整。
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_modify_assay('==0',pseudocount=0.0001,
select_feature = c('1.1.1.102','1.1.1.11'),
select_sample = c('P11774','P31579'))
🏷️示例4:允许使用函数的方式调整数据。
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_modify_assay('>0',pseudocount=function(x) log(x) )

5.7.2 调整微生物物种注释数据
在微生物物种注释数据中经常出现一些丰度极小的菌种,这种情况可能由污染、测序或者数据库比对错误导致。有时需要将这些丰度极小的数据转换为0。
注意:
本示例仅做模块功能展示,如需过滤微生物物种注释数据,请参考5.6 EMP_identify_assay。
本示例仅做模块功能展示,如需过滤微生物物种注释数据,请参考5.6 EMP_identify_assay。
🏷️示例:使用模块EMP_modify_assay将属级别相对丰度小于千分之一的数据转换为0。
调整数据前:
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_collapse(estimate_group = 'Genus',collapse_by='row') |>
EMP_decostand(method = 'relative')

调整后数据:
